Baja variabilidad de ADN satelital en poblaciones naturales de Drosophila antonietae involucradas en diferentes eventos evolutivos
Traducción automática
El artículo original está escrito en idioma EN (enlace para leerlo) .
Drosophila antonietae es una especie cactofílica que se encuentra en el bosque mesofílico de la cuenca del río Paraná–Paraguay y en las dunas de la costa atlántica sur de Brasil. Aunque la estructura genética de las poblaciones de la cuenca del río Paraná–Paraguay ya ha sido establecida, la relación entre estas poblaciones y las de la costa atlántica es controvertida. En este estudio, comparamos 33 unidades repetitivas de ADN satélite pBuM-2 aisladas de individuos de 8 poblaciones de D. antonietae en estas regiones geográficas, incluyendo algunas poblaciones encontradas dentro de una zona de contacto con la estrechamente relacionada D. serido. Las secuencias de pBuM-2 mostraron baja variabilidad interpopulacional. Este resultado se interpretó como consecuencia tanto del flujo génico entre las poblaciones como del cruce desigual que promueve la homogeneización de los arreglos en tándem. Los resultados presentados aquí, junto con los de estudios previos, destacan el uso de pBuM-2 para resolver conflictos taxonómicos dentro del grupo de especies D. buzzatii.
Introducción
Drosophila antonietae es un miembro del grupo de especies D. buzzatii, un grupo monofilético compuesto por 7 especies hermanas que son endémicas de forma natural en la región neotropical, que se encuentran en los bosques abiertos y secos del este de América del Sur (Manfrin y Sene 2006). Estas especies utilizan tejido de cactus necrótico como sitios de cría (Pereira et al. 1983); debido a esto, su rango geográfico está restringido al rango de distribución de los cactus hospedadores. Así, D. antonietae es un modelo atractivo para estudiar procesos evolutivos (Manfrin y Sene 2006).
La distribución geográfica de D. antonietae se puede caracterizar de 2 maneras. En los valles encerrados por la cuenca del río Paraná-Paraguay, se encuentran poblaciones en bosques mesofílicos asociados con el cactus Cereus hildemannianus (Tidon-Sklorz y Sene 2001; Mateus y Sene 2003). En su distribución sureste, D. antonietae se encuentra en las regiones de dunas de la costa atlántica, utilizando principalmente C. hildemannianus pero también cactáceas del género Opuntia (Ruiz et al. 2000; Manfrin y Sene 2006) (Figura 1).
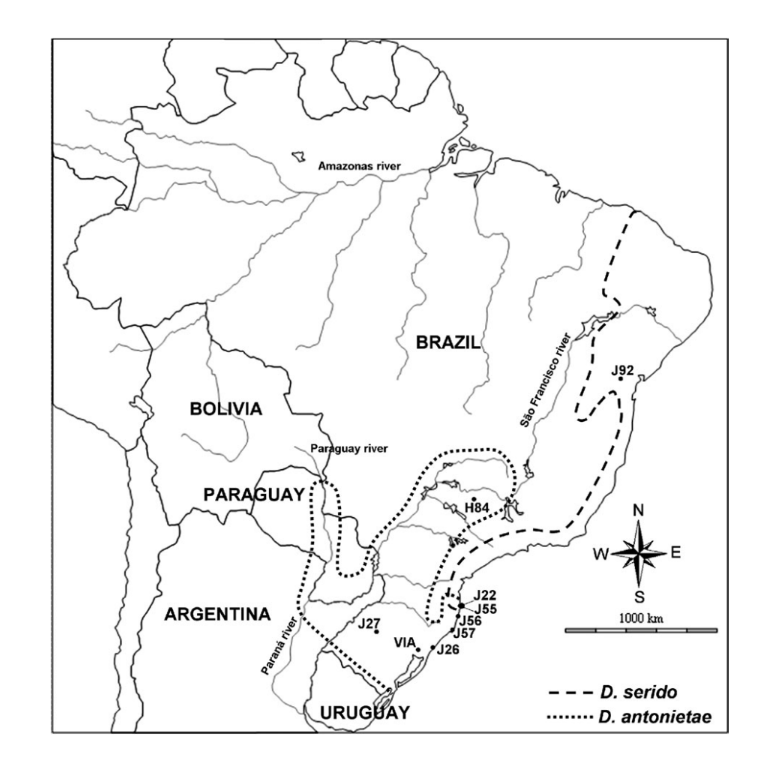
Estudios previos han sugerido que las poblaciones de D. antonietae de la cuenca del río Paraná-Paraguay están estructuradas de acuerdo con el modelo de aislamiento por distancia debido a la correlación positiva de las distancias morfológicas y genéticas con las distancias geográficas y ecológicas (esta última definida como la distancia entre 2 poblaciones siguiendo el curso del río) (de Brito et al. 2002; Mateus y Sene 2007). Basado en el número de migrantes por generación, Mateus y Sene (2007) sugirieron un efecto moderado tanto del flujo génico como de la deriva genética en estas poblaciones de D. antonietae, con el flujo génico superponiéndose a la deriva genética promoviendo un gradiente genético.
Un análisis filogeográfico basado en ADN mitocondrial sugirió que la distribución sureste de D. antonietae, en la costa atlántica de Brasil, fue colonizada por eventos de expansión de poblaciones de D. antonietae de la depresión central del estado de Rio Grande do Sul (de Brito et al. 2002; Manfrin y Sene 2006). Sin embargo, este análisis no pudo determinar si esta expansión fue seguida por el mantenimiento del flujo génico, con adquisición de estructura poblacional de acuerdo con el modelo de aislamiento por distancia, o seguida por fragmentación, implicando una ausencia de flujo génico entre las poblaciones de la costa atlántica y las de la cuenca del río Paraná-Paraguay (de Brito et al. 2002). Los datos de inversión cromosómica podrían sugerir algún grado de fragmentación entre las poblaciones de estos 2 ambientes diferentes, ya que aquellas poblaciones de los valles de la cuenca del río Paraná-Paraguay son polimórficas para las inversiones 2y8, 2z8 y 5e, mientras que las poblaciones de la costa atlántica son polimórficas para 2z8 (Ruiz et al. 2000). Las poblaciones de D. antonietae de la costa atlántica tienen otra característica importante: están en contacto con la especie estrechamente relacionada D. serido (D. buzzatii grupo) (Ruiz et al. 2000; Manfrin y Sene 2006). Esta área de superposición es el resultado de D. serido expandiéndose desde el norte hacia la costa atlántica sur y compartiendo un lugar geográfico con
D. antonietae poblaciones (de Brito et al. 2002; Manfrin y Sene 2006) (Figura 1). Rasgos morfológicos y análisis de ADN mitocondrial sugieren que ha ocurrido hibridación entre
D. antonietae y D. serido dentro de la zona de contacto (C.K.B. Santos y colegas, datos no publicados).
Los ADN satélites son secuencias de ADN altamente repetitivas dispuestas en tándem que representan un componente sustancial de la heterocromatina de organismos eucariotas (Charlesworth et al. 1tt4; Palomeque y Lorite 2008). Los satélites se localizan típicamente cerca de regiones centroméricas y, con menos frecuencia, en telómeros (Ugarkovic y Plohl 2002; Kuhn et al. 2008). Estas secuencias evolucionan de manera concertada, mientras que los principales mecanismos moleculares involucrados en su evolución son la replicación por deslizamiento, el cruce desigual, la conversión de genes y la replicación en círculo rodante (Dover 1982; Charlesworth et al. 1994).
Algunas de las familias de ADN satélite evolucionan rápidamente, lo que permite su uso como marcadores moleculares en estudios comparativos interespecíficos (Watabe et al. 1997; Picariello et al. 2002; Kuhn y Sene 2005). Sin embargo, el uso de estas secuencias como marcadores moleculares en estudios de población ha sido relativamente poco explorado, a pesar de que se informó de evolución concertada en poblaciones de Cyprinodon variegates (Cyprinodontidae) (Elder y Turner 1994) y Acrossocheilus paradoxus (Cyprinidae) (Wu et al. 1999).
Tres familias de ADN satélite compartidas entre las especies del grupo D. buzzatii han sido descritas formalmente: pBuM (Kuhn et al. 1999, 2008; Kuhn y Sene 2005), DBC-150 (Kuhn et al. 2007), y SSS139 (Franco et al. 2008). La familia pBuM está compuesta por 2 subfamilias, pBuM-1 y pBuM-2 (Kuhn y Sene 2005; Kuhn et al. 2008). La subfamilia pBuM-2 está compuesta por monómeros ricos en AT de 370 pb, que presentan diferencias cualitativas (sustitución de nucleótidos fijos) y cuantitativas (número de copias) entre las especies del grupo D. buzzatii (Kuhn y Sene 2005; Kuhn et al. 2007, 2008).
En este trabajo, demostramos la variabilidad intraespecífica del ADN satélite pBuM-2 en D. antonietae, utilizando poblaciones de 2 ambientes diferentes (la cuenca del Paraná–Paraguay y la costa atlántica) que presentan historias evolutivas distintas, asociadas con el mantenimiento del flujo genético en la primera y eventos de hibridación con la especie estrechamente relacionada D. serido en la segunda. Elegimos utilizar el ADN satélite pBuM como un marcador molecular porque exhibe 14 sustituciones nucleotídicas diagnósticas entre D. antonietae y D. serido (Kuhn y Sene 2005). Así, pBuM permite la identificación taxonómica de especímenes aislados de la zona de contacto. Además, dado que la dinámica evolutiva de las secuencias de ADN satélite a nivel intraespecífico no se comprende completamente, datos empíricos adicionales podrían contribuir a una mejor comprensión del proceso subyacente a la evolución del ADN satélite (Ugarkovic y Plohl 2002; Palomeque y Lorite 2008).
Material y Métodos
Muestras
Estudiamos 8 poblaciones brasileñas de D. antonietae (Figura 1). Tres de estas poblaciones se encuentran en los valles de la cuenca del río Paraná–Paraguay: J2/ (Santiago, estado de Rio Grande do Sul), Via (Viamão, estado de Rio Grande do Sul) y H84 (Serrana, estado de São Paulo, número de acceso AY656609– AY656615; Kuhn y Sene 2005); y 5 se encuentran en las dunas de la costa atlántica, dentro de la zona de contacto con
D. serido: J56 (Garopaba, estado de Santa Catarina), J55 (playa Armaxcão, Florianópolis, estado de Santa Catarina), J22 (playa Joaquina, Florianópolis, estado de Santa Catarina), J5/ (Laguna, estado de Santa Catarina) y J26 (Osório, estado de Rio Grande do Sul) (Figura 1). Las secuencias de D. antonietae fueron analizadas junto con las secuencias de G pBuM-2 de D. serido (población Jf2—Milagres, estado de Bahia, número de acceso AY656616–AY656621; Kuhn y Sene 2005), con el fin de identificar algunos eventos de introgresión en poblaciones de la costa atlántica.
Aislamiento del ADN satélite pBuM-2 y análisis de secuencia
El ADN genómico de un individuo macho (moscas capturadas en la naturaleza) por población fue extraído utilizando el Kit de Purificación de ADN Genómico Wizard (Promega). El ADN genómico se sometió a reacción en cadena de la polimerasa para amplificar las copias de la familia pBuM-2 [cebadores A2F (CGGAGTA-TTTTTCATTCGAC) y A2R (GGTATGCCATAAAG-AAGTCG)] de acuerdo con Kuhn et al. (2008). Las bandas resultantes de aproximadamente 400 bp se eluyeron del gel mediante incubación nocturna en una solución de elución (500 mM NaAc; 1 mM ácido etilenodiaminotetraacético). Los fragmentos recuperados se clonaron utilizando el kit de clonación de extremos romos pMOSBlue (RPN 5110; Amersham Pharmacia Biotech). El ADN plasmídico se preparó siguiendo la metodología descrita en Sambrook et al. (1989), y la reacción de plantilla de ADN para secuenciación se preparó de acuerdo con el manual del Kit de Reacción Lista para Secuenciación BigDye Terminator (PerkinElmer). La secuenciación automática de ADN se realizó en un secuenciador ABI Prism 377 (PerkinElmer).
Se realizaron alineamientos de secuencias en CLUSTALW 1.8 (Thompson et al. 1994) y se editaron en BioEdit (Hall 1999). Utilizamos el algoritmo y el modelo estadístico propuesto por Betrán et al. (1997) para detectar tramos de conversión génica. El parámetro Rm, que estima el número mínimo de eventos de recombinación en una muestra de ADN (Hudson y Kaplan 1985), se utilizó para detectar eventos de recombinación. Aunque este parámetro no mide específicamente el cruce desigual, es razonable suponer que el Rm tendría valores superiores a cero si se hubiera producido un cruce desigual. En este sentido, utilizamos Rm como evidencia indirecta de cruce desigual. Las distancias genéticas se calcularon de acuerdo con p-distancia, que es la proporción de diferencias nucleotídicas entre 2 secuencias (es decir, p 5 Np/N, donde Np son las diferencias de nucleótidos y N es el número total de nucleótidos). También se aplicaron modelos de distancia más complejos a los datos, con resultados similares a los presentados por p-distancia (datos no mostrados). La matriz de distancias se utilizó para crear un fenograma utilizando el método de vecino más cercano (Saitou y Nei 1987). Los cálculos de distancia genética y los fenogramas se realizaron utilizando el programa MEGA 3.0 (Kumar et al. 2004).
También investigamos patrones de variación intraespecífica en D. antonietae mediante análisis de varianza molecular (AMOVAs; Excoffier et al. 1992). Para el AMOVA, las poblaciones de D. antonietae se agruparon en 2 grupos: costa atlántica y cuenca del río Paraná-Paraguay. El AMOVA se realizó utilizando Arlequin 3.0 (Excoffier et al. 2005).
Resultados y Discusión
Se obtuvieron veintiséis nuevos monómeros de ADN satélite pBuM-2: 4 de la localidad J56 (número de acceso FJ935973– FJ93597G); 4 de la localidad Via (número de acceso FJt35t77–FJt35t80); 4 de la localidad J26 (número de acceso FJ935981–FJ935984); 3 de la localidad J5/ (número de acceso FJ935985–FJ935987); 5 de la localidad J2/ (número de acceso FJ935988–FJ935992); 2 de la localidad J55 (número de acceso FJ935993–FJ935994); y 4 de la localidad J22 (número de acceso FJ935995–FJ935998). La alta similitud de nucleótidos entre las secuencias obtenidas en este trabajo y las descritas previamente (Kuhn y Sene 2005) confirma que pertenecen a la familia de ADN satélite pBuM-2.
Todas las secuencias obtenidas presentan la estructura primaria típica de D. antonietae y se agruparon en la misma rama en el fenograma, junto con las secuencias pBuM-2 de D. antonietae previamente descritas (Figura 2), incluso aquellas de las poblaciones dentro de la zona de contacto con D. serido, donde se sugirió hibridación entre D. antonietae y D. serido con introgressión de ADN mitocondrial (C.K.B Santos, F.M. Sene y M.H. Manfrin, datos no publicados). Esta situación podría estar relacionada con las observaciones de que los genes mitocondriales tienden a introgressar más fácilmente que las secuencias nucleares (Dorado et al. 1992; Arnold 1993). Las explicaciones más citadas para esta observación están relacionadas con procesos de desequilibrio cito-nuclear (Arnold 1993) y la esterilidad de los machos híbridos (Aubert y Solignac 1990).
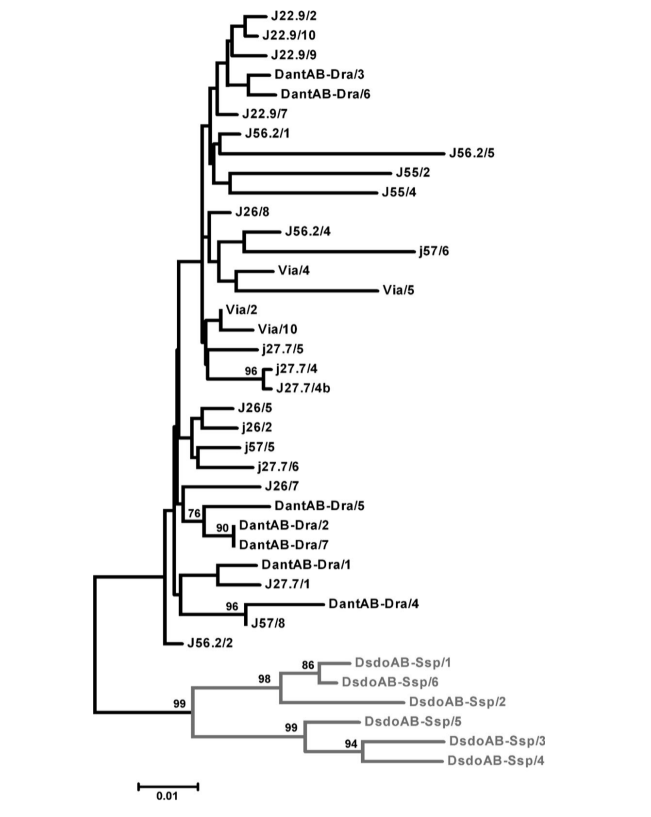
Hay un alto nivel de similitud de secuencia entre las secuencias de pBuM-2 de D. antonietae (95.7% en promedio), y no se encontraron mutaciones que fueran diagnósticas para una población particular. La variabilidad interpopulacional fue muy similar a la variabilidad intrapopulacional (Tabla 1), y las secuencias de la misma población no se agruparon en una rama específica en el dendrograma (Figura 2). La AMOVA realizada con las poblaciones subdivididas por grupos geográficos (cuenca del río Paraná-Paraguay y costa atlántica) mostró que el 99.30% de la diversidad genética total podría atribuirse a la variabilidad intragrupo, mientras que solo el 0.70% se atribuyó a la variabilidad intergrupo (Tabla 2). El Фst calculado fue bajo y no tuvo soporte estadístico (Tabla 2).
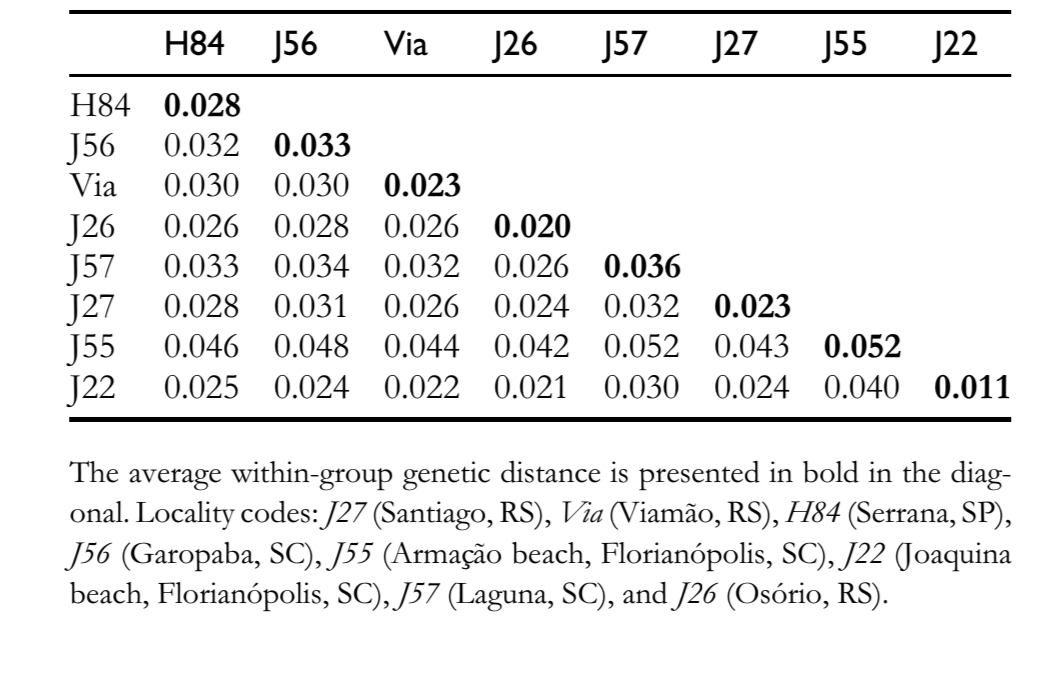

Las poblaciones de D. antonietae de la cuenca del río Paraná-Paraguay están estructuradas según un modelo de aislamiento por distancia (Monteiro y Sene 1995; Mateus y Sene 2007). Las inversiones cromosómicas observadas sugieren fragmentación entre las poblaciones de la costa atlántica y las de la cuenca del río Paraná-Paraguay (Ruiz et al. 2000), mientras que los datos de ADN mitocondrial no pudieron concluir si la fragmentación o el aislamiento por distancia era la estructura dominante que definía la relación de estos 2 grupos de población (de Brito et al. 2002). La alta similitud nucleotídica de pBuM-2 en D. antonietae (Figura 2 y Tablas 1 y 2) es congruente con el mantenimiento del flujo génico entre las poblaciones de esta especie, incluso entre las asignadas en la cuenca del río Paraná-Paraguay y la costa atlántica. Nuestros resultados sugieren que el aislamiento por distancia debería explicar la variabilidad del ADN mitocondrial en D. antonietae; este modelo predice flujo génico entre las poblaciones, como se infiere a partir de los datos de ADN satelital presentes aquí. En este contexto, los menores polimorfismos de inversión cromosómica en las poblaciones de la costa atlántica de D. antonietae, detectados por Ruiz et al. (2000), podrían explicarse como consecuencia de eventos históricos y demográficos, como la expansión poblacional desde las poblaciones de los valles de la cuenca del río Paraná-Paraguay, como lo sugiere el ADN mitocondrial (de Brito et al. 2002). Alternativamente, esta diferenciación poblacional podría estar asociada a la adaptación local a un entorno distinto en presencia de flujo génico, ya que las inversiones cromosómicas pueden estar correlacionadas con características adaptativas en Drosophila cactofílica (Hasson et al. 1992; Fernández Iriarte et al. 2003). Una explicación alternativa para la alta conservación nucleotídica de pBuM-2 en D. antonietae se basa en la dinámica evolutiva de las secuencias de ADN satelital. Los mecanismos moleculares relacionados con la homogeneización de secuencias dispuestas en tándem, como el cruce desigual y la conversión génica a través de un proceso llamado impulso molecular (Dover 1982; Strachan et al. 1985), podrían estar actuando sobre el ADN satelital pBuM en esta especie. Nuestras análisis no identificaron tramos de conversión génica en las secuencias de pBuM-2 aisladas de D. antonietae, sugiriendo que este mecanismo molecular no influye en la diversidad de secuencias de pBuM-2 en esta especie. Sin embargo, se detectó un mínimo de 4 eventos de recombinación en la muestra (Rm = 4) entre los intervalos de 130–136, 136–155, 174–376 y 376–378. Estos eventos de recombinación podrían ser una evidencia indirecta de cruce desigual que contribuye al mantenimiento de la estructura primaria de las secuencias de pBuM-2 en las diferentes poblaciones de D. antonietae. Consistente con esta hipótesis, el genoma de D. antonietae contiene un gran número de monómeros de pBuM-2 (Kuhn et al. 2007; Kuhn et al. 2008), un hecho que aumenta la probabilidad de cruce desigual (Smith1976), y, en consecuencia, la homogeneización y evolución concertada.
Estas 2 hipótesis alternativas que explican la alta similitud de nucleótidos encontrada en los monómeros de pBuM-2 de D. antonietae no son mutuamente excluyentes. En este contexto, proponemos que un equilibrio entre el flujo genético entre poblaciones y los eventos de homogeneización de los arreglos en tándem de ADN satélite, promovidos por el entrecruzamiento desigual, contribuye a la conservación de la estructura primaria de pBuM-2 en las especies de D. antonietae.
La ausencia de divergencia molecular en las secuencias de ADN satélite entre poblaciones de la misma especie, como se encontró en el presente artículo, también se ha observado en otras especies de insectos (Bachmann et al. 1994, 1998; Lorite et al. 2002; Feliciello et al. 2005). Por ejemplo, Bachmann et al. (1994) encontraron altos niveles de similitud genética entre los monómeros del pDoP102 satélite en poblaciones de Dolichopoda schiavazzii (Orthoptera), cuyos datos alozimáticos indican que han estado aislados entre sí durante al menos 235 000 años. Según estos autores, la homogeneidad en pDoP102 incluso en poblaciones aisladas podría deberse a la biología del ADN satélite porque el entrecruzamiento desigual y la conversión génica pueden inducir el mantenimiento de la estructura primaria de las secuencias de ADN satélite (Bachmann et al. 1994). En otro estudio, la presencia de tramos de conversión génica en los monómeros de una familia de ADN satélite se utilizó como evidencia de eventos de homogeneización en este ADN satélite, explicando la alta conservación intraspecífica de ADN satélite encontrada en el escarabajo fitófago Xanthogaleruca luteola (Lorite et al. 2002).
En la especie del grupo D. buzzatii , también se identificó una alta conservación intraespecífica de la secuencia pBuM-2 en D. seriema (Kuhn y Sene 2004) y D. gouveai (Franco, Kuhn, et al. 2006). Además, las poblaciones de D. buzzatii son homogéneas en lo que respecta al ADN satélite pBuM-1, otro miembro de la familia pBuM (Kuhn et al. 2003). En D. seriema y D. buzzatii, se sugirió que la falta de diferenciación poblacional en las secuencias de ADN satélite podría deberse a eventos de flujo génico pasados y actuales entre poblaciones, que son compatibles con los resultados obtenidos de otros marcadores genéticos analizados en las mismas poblaciones (Kuhn et al. 2003; Kuhn y Sene 2004). Para
D. gouveai, que está estructurado según un modelo de ‘‘isla’’ (de Brito et al. 2002; Moraes y Sene 2007), la hipótesis principal para explicar la conservación de la estructura primaria en el ADN satélite pBuM-2 fue que ocurrieron mecanismos de homogenización independientes que favorecen unidades repetitivas compartidas entre las poblaciones después del evento de aislamiento de las poblaciones de D. gouveai (Franco, Kuhn, et al. 2006). El clúster de D. buzzatii está compuesto por especies hermanas con una taxonomía basada en las características morfométricas del aedeago, que es el órgano intromitente de los genitales masculinos de los insectos (Vilela 1983; Tidon-Sklorz y Sene 2001; Franco, Prado, et al. 2006). Así, la identificación de individuos hembras del clúster de D. buzzatii, proporcionados del entorno natural, se realiza mediante la identificación de su descendencia masculina, lo que hace necesario el establecimiento de líneas isofemeninas, lo cual puede ser una tarea ardua para las especies de Drosophila cactofílicas. Además, la correcta discriminación de especies basada solo en caracteres cuantitativos, como la morfología del aedeago, a veces es difícil debido a la presencia de individuos morfológicamente ambiguos, especialmente cuando los individuos son recolectados dentro de zonas de contacto entre especies del clúster de D. buzzatii. En este sentido, la fuerte conservación intraespecífica de las secuencias de ADN satélite pBuM obtenidas en varios estudios (Kuhn et al. 2003; Kuhn y Sene 2004; Franco, Kuhn, et al. 2006; documento presente), incluso en especies estructuradas genéticamente, destaca la utilidad de pBuM-2 para resolver conflictos taxonómicos dentro de las especies del clúster de D. buzzatii, un modelo reconocido para estudios de biología evolutiva (Manfrin y Sene 2006).
Autores: Fernando Faria Franco, Fabio Melo Sene, Maura Helena Manfrin
Referencias:
- Arnold J. 1993. Desequilibrios cito-nucleares en zonas híbridas. Annu Rev Ecol Syst. 24:521–554.
- Aubert J, Solignac M. 1990. Evidencia experimental de la introgresión de ADN mitocondrial entre especies de Drosophila. Evolution. 44:1272–1282.
- Bachmann L, Tomiuk J, Adis J, Vohland K. 1998. Diferenciación genética del milpiés Pycnotropis epiclysmus que habita en bosques amazónicos inundados estacionalmente y no inundados. J Zool Syst Evol Res. 36:65–70.
- Bachmann L, Venanzetti F, Sbordoni V. 1994. Caracterización de una familia de ADN satélite específica de especie de Dolichopoda schiavazzi (Orthoptera, Rhaphidophoridae) grillos de cueva. J Mol Evol. 39:274–281.
- Betrán E, Rozas J, Navarro A, Barbadilla A. 1997. La estimación del número y la distribución de longitud de los tramos de conversión genética a partir de datos de secuencia de ADN poblacional. Genetics. 146:89–99.
- Charlesworth B, Sniegowski P, Stephan W. 1tt4. La dinámica evolutiva del ADN repetitivo en eucariotas. Nature. 371:215–220.
- de Brito AR, Manfrin MH, Sene FM. 2002. Análisis cladístico anidado de poblaciones brasileñas de Drosophila serido. Mol Phylogenet Evol. 22: 131–143.
- Dorado O, Rieseberg LH, Arias D. 1992. Introgresión de ADN cloroplástico en girasoles del sur de California. Evolution. 46:566–572.
- Dover G. 1982. Impulso molecular: un modo cohesivo de evolución de especies. Nature. 299:111–117.
- Elder JF, Turner BJ. 1tt4. Evolución concertada a nivel poblacional: secuencias de ADN satélite HindIII de peces de aleta. Proc Natl Acad Sci U S A. 91: 994–998.
- Excoffier L, Laval G, Schneider S. 2005. Arlequin (versión 3.0): un paquete de software integrado para el análisis de datos de genética de poblaciones. Evol Bioinform Online. 1:47–50.
- Excoffier L, Smouse PE, Quattro JM. 1992. Análisis de la varianza molecular inferida a partir de distancias métricas entre haplotipos de ADN: aplicación a datos de restricción de ADN mitocondrial humano. Genetics. 131:479–491.
- Feliciello I, Picariello O, Chinali G. 2005. La primera caracterización de la variabilidad general de unidades repetitivas en una especie revela características inesperadas del ADN satélite. Gene. 349:153–164.
- Fernández Iriarte PJ, Norry FM, Hasson ER. 2003. Inversiones cromosómicas afectan el tamaño y la forma del cuerpo en diferentes recursos de cría en Drosophila buzzatii. Heredity. 91:51–59.
- Franco FF, Kuhn GCS, Sene FM, Manfrin MH. 2006. Conservación de secuencias de ADN satélite pBuM–2 entre poblaciones geográficamente aisladas de Drosophila gouveai de Brasil. Genetica. 128:287–295.
- Franco FF, Prado PRR, Sene FM, Costa LF, Manfrin MH. 200G. Morfología del aedeago como marcador discriminante en dos especies cactofílicas estrechamente relacionadas de Drosophila (Diptera; Drosophilidae) en América del Sur. An Acad Bras Cienc. 78:203–212.
- Franco FF, Sene FM, Manfrin MH. 2008. Caracterización molecular de la familia de ADN satélite SSS13t en especies hermanas del grupo Drosophila buzzatii. Genet Mol Biol. 31:155–159.
- Hall TA. 1999. BioEdit: un editor de alineación de secuencias biológicas fácil de usar y programa de análisis para Windows t5/t8/NT. Nucleic Acids Symp Ser. 41:95–98.
- Hasson E, Fanara JJ, Rodríguez C, Vilardi JC, Reig OA, Fontdevila A. 1992. La historia evolutiva de Drosophila buzzatii. XXIV. Las inversiones del segundo cromosoma tienen diferentes efectos promedio sobre la longitud del tórax. Heredity. 68:557–563.
- Hudson RR, Kaplan NL. 1985. Propiedades estadísticas del número de eventos de recombinación en la historia de una muestra de secuencias de ADN. Genetics. 111:147–164.
- Kuhn GCS, Bollgönn S, Sperlich D, Bachmann L. 1999. Caracterización de un ADN satélite específico de especie de Drosophila buzzatii. J Zool Syst Evol Res. 37:109–112.
- Kuhn GCS, Franco FF, Manfrin MH, Moreira-Filho O, Sene FM. 2007. Bajas tasas de homogeneización de la familia de ADN satélite DBC-150 restringida a un solo par de microcromosomas en especies del grupo Drosophila buzzatii. Chromosome Res. 15:457–469.
- Kuhn GCS, Franco FF, Silva WA Jr, Martinez-Rossi NM, Sene FM. 2003. Sobre la variabilidad del ADN satélite pBuM18t entre poblaciones sudamericanas de Drosophila buzzatii. Hereditas. 139:161–166.
- Kuhn GCS, Sene FM. 2004. Caracterización y variabilidad inter-poblacional de un ADN satélite complejo HpaI de Drosophila seriema (grupo repleta). Genetica. 121:241–249.
- Kuhn GCS, Sene FM. 2005. Rotación evolutiva de dos subfamilias de ADN satélite pBuM en el grupo de especies Drosophila buzzatii (grupo repleta): de alpha a alpha/beta arreglos. Gene. 349:77–85.
- Kuhn GCS, Sene FM, Moreira-Filho O, Schwarzacher T, Heslop-Harrison JS. 2008. El análisis de secuencias, distribución cromosómica y organización a largo alcance muestran que la rápida rotación de repeticiones de ADN satélite pBuM nuevas y viejas conduce a diferentes patrones de variación en siete especies del grupo Drosophila buzzatii. Chromosome Res. 16:307–324.
- Kumar S, Tamura K, Nei M. 2004. MEGA3: software integrado para análisis de genética evolutiva molecular y alineación de secuencias. Brief Bioinform. 5:150–163.
- Lorite P, Carrillo JA, Garneria I, Petitpierre E, Palomeque T. 2002. ADN satélite en el escarabajo de la hoja de olmo, Xanthogaleruca luteola (Coleoptera, Chrysomelidae): caracterización, análisis inter-poblacional y ubicación cromosómica. Cytogenet Genome Res. 98:302–307.
- Manfrin MH, Sene FM. 200G. Drosophila cactofílica en América del Sur: un modelo para estudios evolutivos. Genetica. 126:57–75.
- Mateus RP, Sene FM. 2003. Variación alozima temporal y espacial en la Drosophila antonietae cactofílica sudamericana (Diptera; Drosophilidae). Biochem Genet. 41:219–233.
- Mateus RP, Sene FM. 2007. Estudio genético poblacional de la variación alozima en poblaciones naturales de Drosophila antonietae (Insecta, Diptera). J Zool Syst Evol Res. 45:136–143.
- Monteiro SG, Sene FM. 1tt5. Estudio morfométrico de poblaciones de Drosophila serido de las regiones Central y Sur de Brasil. Rev Bras Genet (Suppl). 18:283.
- Moraes EM, Sene FM. 2007. Variación en microsatélites y morfometría en Drosophila gouveai: la importancia relativa de factores históricos y actuales en la configuración de la estructura genética poblacional. J Zoolog Syst Evol Res. 45:336–344.
- Palomeque T, Lorite P. 2008. ADN satélite en insectos: una revisión. Heredity. 100:564–573.
- Pereira MAQR, Vilela CR, Sene FM. 1983. Notas sobre sitios de cría y alimentación de algunas especies del grupo repleta del género Drosophila (Diptera, Drosophilidae). Cienc Cult. 35:1313–1319.
- Picariello O, Feliciello I, Bellinero R, Chinali G. 2002. ADN satélite S1 como marcador taxonómico en ranas marrones: evidencia molecular de que Rana graeca graeca y Rana graeca italica son especies diferentes. Genome. 45: 63–70.
- Ruiz A, Cassian AM, Kuhn GCS, Alves MAR, Sene FM. 2000. El rompecabezas de especiación de Drosophila serido: juntando nuevas piezas. Genetica. 108:217–227.
- Saitou N, Nei M. 1987. El método de vecino-uniendo: un nuevo método para reconstruir árboles filogenéticos. Mol Biol Evol. 4:406–425.
- Sambrook J, Fritsh EF, Maniatis T. 1989. Clonación molecular: un manual de laboratorio. 2ª ed. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press.
- Smith GP. 1976. Evolución de secuencias de ADN repetidas por cruce desigual. Science. 191:528–535.
- Strachan T, Webb D, Dover GA. 1985. Etapas de transición del impulso molecular en familias de ADN de múltiples copias en Drosophila. EMBO J. 4:1701–1708.
- Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: mejorando la sensibilidad de la alineación progresiva de múltiples secuencias a través de ponderación de secuencias, penalizaciones de huecos específicas de posiciones y elección de matriz de pesos. Nucleic Acids Res. 22:4673–4680.
- Tidon-Sklorz R, Sene FM. 2001. Dos nuevas especies del conjunto de hermanos de Drosophila serido (Diptera, Drosophilidae). Iheringia. 90:141–146.
- Ugarkovic D, Plohl M. 2002. Variación en perfiles de ADN satélite: causas y efectos. EMBO J. 21:5955–5959.
- Vilela CR. 1983. Una revisión del grupo de especies Drosophila repleta (Diptera; Drosophilidae). Rev Bras Entomol. 27:1–114.
- Watabe H, Bachmann L, Haring E, Sperlich D. 1997. Estudios taxonómicos y moleculares sobre Drosophila sinobscura y D. hubeiensis, dos especies hermanas del grupo D. obscura. J Zool Syst Evol Res. 35:81–94.
- Wu WL, Wang JP, Tseng MC, Chiang TY. 1999. Clonación y variabilidad genética de un ADN repetitivo HindIII en Acrossocheilus paradoxos (Cyprinidae). Genome. 42:780–788.
