Variación Genética en Drosófilas de Hawái. IV. Similitud Alozímica Entre D. Silvestris y D. Heteroneura de la Isla de Hawái
Traducción automática
El artículo original está escrito en idioma EN (enlace para leerlo) .
Resumen
Las especies son endémicas de la isla más nueva del archipiélago y son ampliamente simpátricas. Se distinguen fácilmente morfológicamente en ambos sexos. Utilizando procedimientos electroforéticos estándar, hemos examinado 25 loci que codifican para proteínas estructurales de 539 silvestris y 325 heteroneura recogidos en tres localidades ampliamente separadas donde las dos especies son simpátricas. Las comparaciones por pares dentro y entre las especies muestran los siguientes coeficientes de identidad genética (I de Nei): dentro de silvestris 0.961 ± 0.01; dentro de heteroneura 0.949 ± 0.02; entre silvestris y heteroneura, 0.939 ± 0.01. Ni las diferencias dentro ni entre las especies son significativas. No hay diferencias alélicas fijas ni dentro ni entre las especies. En las tres áreas de simpatría, las especies muestran diferencias en la frecuencia de genes P < 0.05) en 9, 11 y 13 loci respectivamente. Esto no es muy diferente de la variación dentro de cualquiera de las especies a través de las tres localidades. Las dos especies tienen niveles similares de heterocigosidad (H) (silvestris, 0.083; heteroneura, 0.089) y porcentaje de loci polimórficos (ambos 0.37). Se sugiere que a pesar de su divergencia morfológica, estas especies están mucho más recientemente formadas que las especies hermanas clásicas. Las diferencias alozímicas significativas pueden no haber tenido tiempo para acumularse.
DROSOPHILA silvestris y D. heteroneura son un par de especies gigantes estrechamente relacionadas endémicas solo de las selvas tropicales de la isla de Hawái, la más nueva del archipiélago. Son simpátricas en la mayoría de las áreas, excepto en altitudes más altas donde las poblaciones tienden a ser exclusivamente silvestris.
Las especies se distinguen fácilmente morfológicamente (Hardy 1965) no solo por la extraordinariamente ancha cabeza en ambos sexos de heteroneura, sino también por los patrones de color de la cara, el tórax, las pleuras y el abdomen, y por las diferencias en el patrón de marcas en las alas. D. silvestris presenta seis inversiones únicas en estado polimórfico; una séptima inversión es polimórfica dentro de ambas especies (Carson y Stalker 1968; Craddock y Carson 1975). No hay inversiones fijas entre las especies, y las dos comparten un conjunto común de secuencias estándar, es decir, son cromosómicamente homosecuenciales.
Aunque hay un fuerte aislamiento sexual entre las especies en cruces de laboratorio (Ahearn et al. 1974; Kaneshiro 1976), se han obtenido híbridos F1 de ambos cruces recíprocos (Craddok 1974; Ahearn y Val 1975). Ambos sexos de estos híbridos F1 son completamente fértiles y se ha observado segregación genética de las diferencias morfológicas de las especies en la generación F1 y en los retrocruces (Val 1976).
Los estudios preliminares de alozimas han mostrado una extraordinaria similitud entre las dos especies (S = 0.96; Johnson et al. 1975; Johnson y Carson 1975). El presente artículo amplía el estudio de la variabilidad genética dentro y entre las dos especies a proteínas solubles codificadas por 25 loci. Los datos provienen de tres áreas de simpatría. Los resultados confirman la notable similitud bioquímica de las especies a pesar de las diferencias genéticas entre ellas en morfología, inversiones y comportamiento.
Materiales y métodos
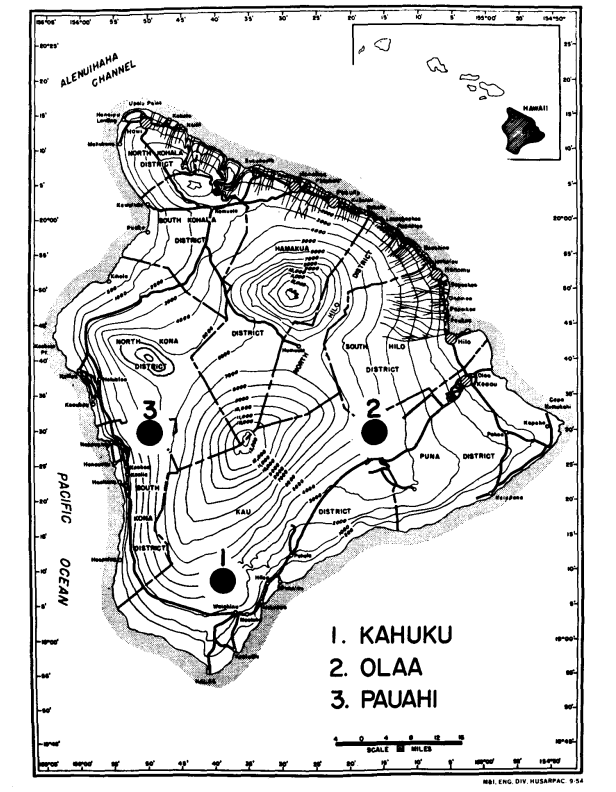
En 1974 y 1975, recolectamos las dos especies de tres áreas de elevación moderada en Hawái (La Gran Isla) donde son simpátricas. Estas son (Figura 1): (1) Kahuku Ranch en la parte sur del Distrito de Kau, (elevación 1158 m); (2) Olaa, bosque cerca de la Estación Experimental del Volcán en Wright Road, Distrito de Puna (elevación 1238 m) y (3) Pauahi, Distrito de Kona Sur (elevación 1311 m). Los machos recién capturados se utilizaron para electroforesis tan pronto como fue posible después de la captura. Las hembras fueron aisladas y se les permitió producir progenie F1 para su uso en preparaciones de glándulas salivales. Después de producir larvas, las madres se utilizaron para electroforesis. Solo el abdomen de las moscas fue sometido a electroforesis; el resto (cabeza, tórax y apéndices) fue montado y colocado en una colección para ser utilizado en análisis morfológicos.
Después de la extracción, el abdomen se congeló durante la noche y luego se homogeneizó individualmente en 0.05 ml de agua destilada desionizada y cada sobrenadante se absorbió mediante cuatro mechas de papel de filtro Whatman No. I (1 X 0.2 cm). Estas mechas se separaron y se aplicaron a cuatro geles de almidón horizontales combinando diferentes sistemas de tampones. Tras la electroforesis, cada gel se cortó horizontalmente cuatro o cinco veces, y estas rebanadas se tiñeron por separado para uno o más de 25 enzimas. Todo el trabajo se realizó utilizando Electrostarch lote 303 a una concentración del 12%.
Las tres combinaciones de tampones utilizadas en el estudio son las siguientes: Sistema de Tampón A = Tampones de gel: pH 8.9, 0.0076 m Tris, 0.005 m citrato; Tampones de electrodos: pH 8.7, 0.269 m borato, 0.1 m hidróxido de sodio. Sistema de Tampón C = Tampones de gel: pH 8.1, 0.074 m Tris, 0.008 m citrato; Tampones de electrodos: pH 8.1, cátodo = 0.343 m Tris, 0.079 m citrato; ánodo = 0.458 m Tris, 0.0104 m citrato. Sistema de Tampón JRP = Tampones de gel: pH 7.0, 0.009 m Tris, 0.003 m citrato; tampones de electrodos: 0.135 m Tris, 0.040 m citrato. Todos estos tampones y los métodos de tinción utilizados son similares a los descritos por Steiner y Johnson (1973) y Ayala et al. (1972; 1974a).
Los alelos en un locus particular fueron numerados de acuerdo con las movilidades relativas de sus alozimas en los sistemas de tampones electroforéticos especificados. El alelo que produce la aloenzima más frecuente fue designado l.00 y los alelos correspondientes a bandas que migran más rápido o más lentamente fueron asignados arbitrariamente valores indicativos de sus respectivas movilidades.
El tampón utilizado y la notación para cada enzima son los siguientes: Sistema de Tampón A : Esterasa (EST, 1 locus) ; Fosfatasa Alcalina (AP, 1 locus) ; Fosfatasa Ácida (ACPH, 1 locus) ; Transaminasa Glutamato Oxaloacetato (GOT, 2 loci) ; Aldehído Oxidasa (AO, 1 locus) ; Alcohol deshidrogenasa (ODH, I locus) ; Deshidrogenasa de Alcohol (ADH, 1 locus) ; Deshidrogenasa de Xantina (XDH, 1 locus) ; Aminopeptidasa de Leucina (LAP, 2 loci) ; Glicerol-3-Fosfato Deshidrogenasa (G-3-PDH, 1 locus). Sistema de Tampón C : Enzima Málica (ME, 1 locus) ; Deshidrogenasa de Malato (MDH, 2 loci) ; Deshidrogenasa de α-Glicerofosfato (α-GPDH, 1 locus) ; Fosfoglucomutasa (PGM, I locus) ; Fumarasa (FUM, 1 locus) ; Deshidrogenasa de Sorbitol (SDH, 1 locus) ; Hexoquinasa (HK, 2 loci) ; Deshidrogenasa de Isocitrato (IDH, 1 locus). Sistema de Tampón JRP : 6-Deshidrogenasa de 6-Fosfogluconato (6-PGDH, 1 locus) ; Deshidrogenasa de Hidroxibutirato (HBDH, 1 locus) ; Isomerasa de Fosfoglucosa (PGI, 1 locus).
Resultados
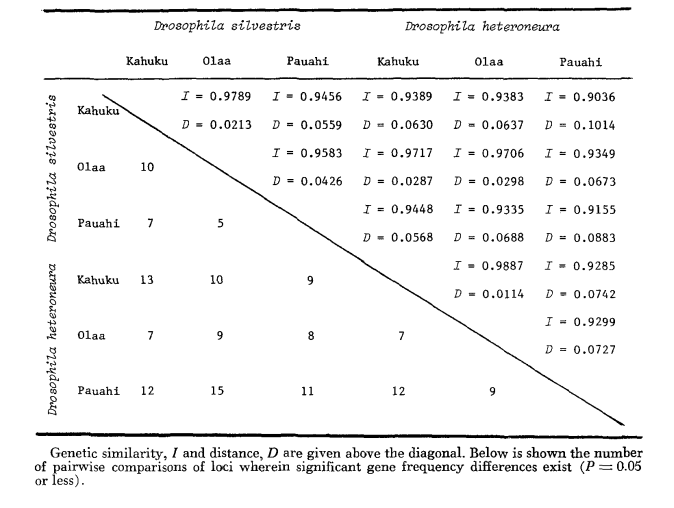
Los datos básicos de alozimas se presentan en la Tabla 1. La Tabla 2 muestra los resultados de usar los datos de la Tabla 1 para calcular coeficientes de similitud genética, I, y distancia, D (Nei 1972) de manera pareada dentro y entre las especies. En la misma tabla (debajo de la diagonal) se muestran el número de loci que presentan diferencias en la frecuencia de genes (P ≤ 0.05) en comparaciones pareadas. El cálculo se realizó, utilizando χ2, usando la frecuencia del alelo más común contra una frecuencia agrupada de los otros alelos. Se utilizó la corrección de Yates.
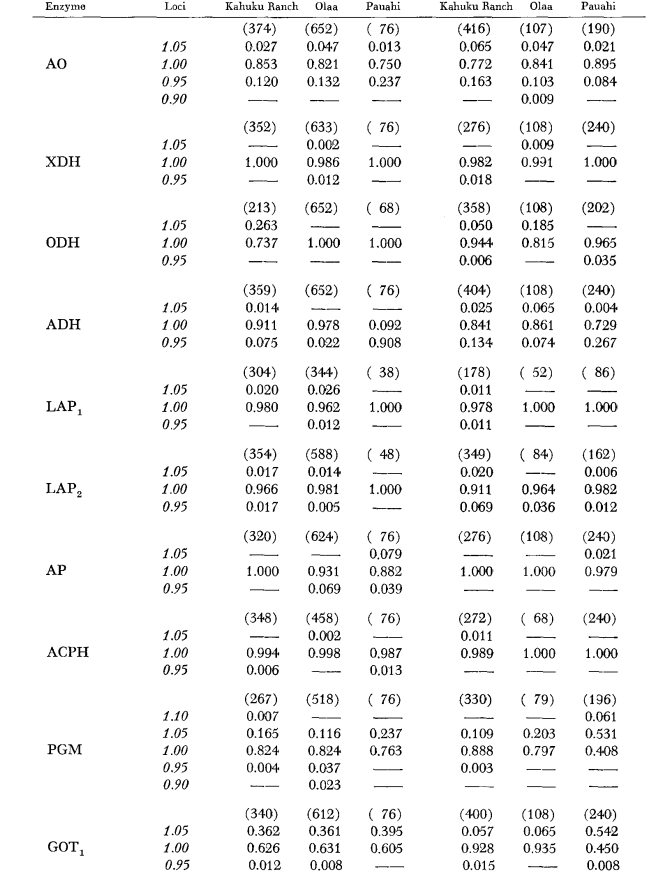
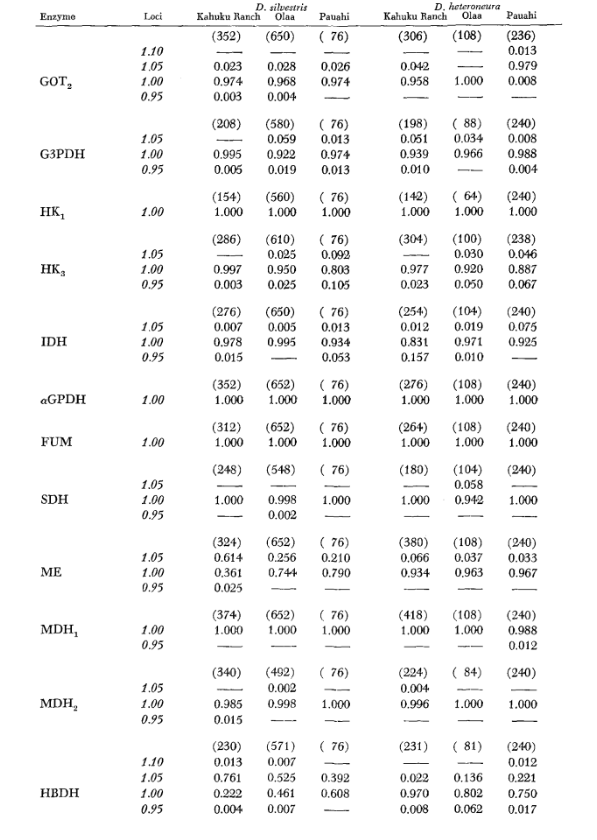
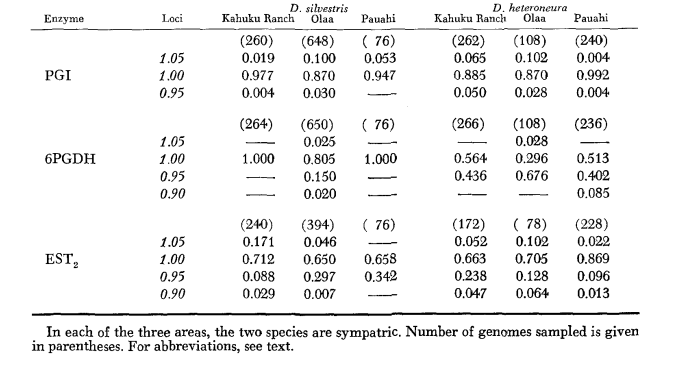
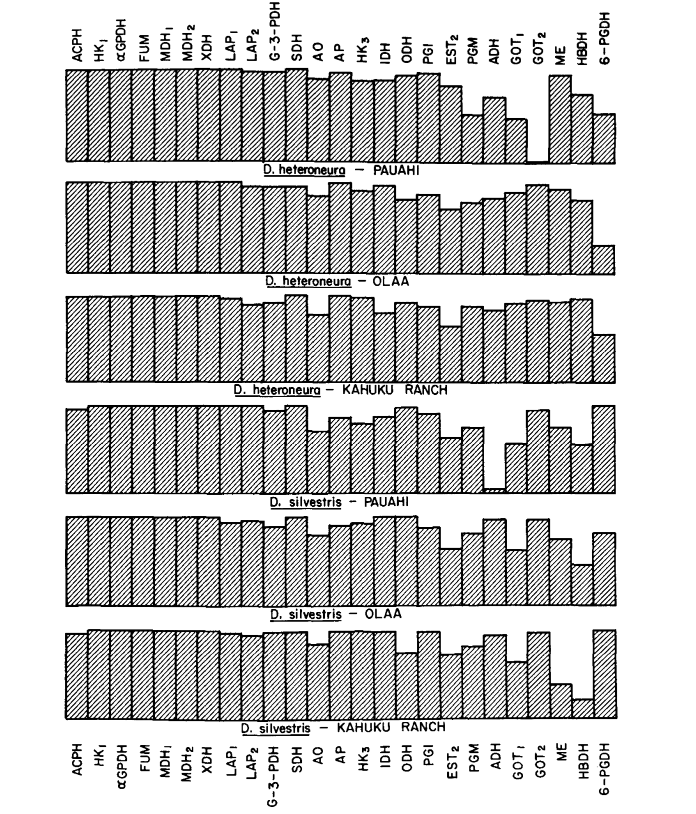
Los niveles promedio de heterocigosidad y las estimaciones de la frecuencia de loci polimórficos se presentan en la Tabla 3. Una representación gráfica de la frecuencia del alelo 1.0 para cada alozima en cada población se presenta en la Figura 2.
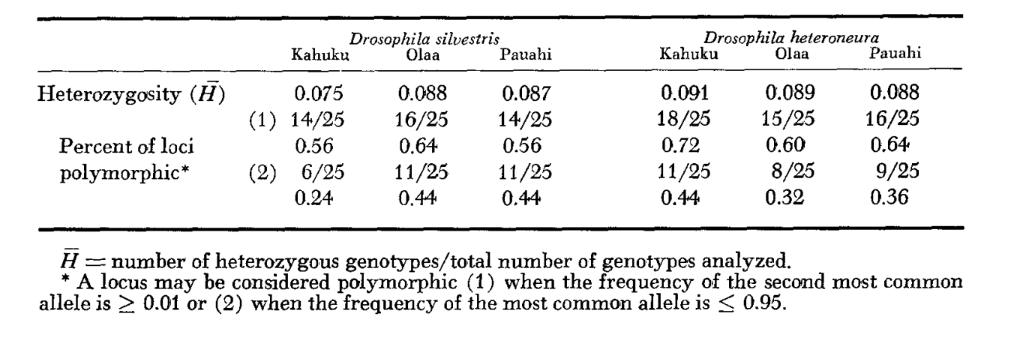
La similitud alozímica es muy grande tanto dentro como entre las especies, ya sea medida por coeficientes de similitud o por diferencias en la frecuencia de genes (ver resumen en la Tabla 4). No hay diferencias alélicas fijas ni dentro ni entre las especies. En las tres áreas de simpatría, las especies muestran diferencias en la frecuencia de genes (P = 0.05) en 9, 11 y 13 loci respectivamente. Esto no es muy diferente de la variación dentro de cualquiera de las especies a través de las tres localidades (silvestris: 5, 7, 10; heteroneura: 7, 9, 12). Las dos especies tienen niveles de heterocigosidad similares (silvestris 0.083; heteroneura 0.089) y porcentaje de loci polimórficos (ambos 0.37).

Algunos detalles son de interés. La similitud entre las especies es menor en Pauahi (Tabla 2; Figura 2). La inspección muestra que esto se debe principalmente a las diferencias de frecuencia en ADH y GOT2. Grandes diferencias en estos loci parecen aparecer entre las especies solo en esta área; es decir, el alelo 1.00 GOT2 es bajo solo en Pauahi heteroneura y el alelo 1.00 de ADH es bajo solo en Pauahi silvestris (Tabla 1; Figura 2).
La similitud entre las especies es algo mayor en el área de Kahuku. Aquí, las diferencias involucran principalmente GOT1, ME y HBDH. En Olaa, la similitud entre las especies es mayor. GOT1 y HBDH nuevamente muestran diferencias, aunque no son tan grandes como las que existen entre las especies en Kahuku.
Discusión
Las comparaciones genéticas de poblaciones utilizando alozimas han inundado la literatura en los últimos diez años (ver reseñas de Ayala 1975 y Powell 1975). Dado que tanto las proteínas estudiadas como los métodos electroforéticos utilizados han sido, en general, similares, los hallazgos de taxones bastante dispares pueden ser comparados de manera útil. Por ejemplo, la similitud genética entre dos especies de lagartos, cuando se reduce a un coeficiente como el de Nei, puede ser comparada directamente con datos similares de pares de especies de ratones, plantas o Drosophila.
En su artículo seminal, Lewontin y Hubby (1966) señalaron una serie de razones por las cuales los métodos electroforéticos estándar estarán sesgados hacia una subestimación de la variabilidad genética y la diferencia genética. Esto debe tenerse en cuenta aún más hoy en día debido al reciente desarrollo del análisis electroforético secuencial. Así, Singh, Lewontin y Felton (1976) y Coyne (1976) han demostrado que, para el locus de la xantina deshidrogenasa de D. pseudo-obscura y persimilis, el número de alelos en muestras naturales bastante pequeñas de estas especies es al menos cuatro veces mayor que el número revelado por los métodos estándar. En lo que respecta a las comparaciones interpopulacionales, estos nuevos métodos parecen seguros para reducir las similitudes observadas en todos los niveles de diferenciación poblacional (por ejemplo, poblaciones locales, subespecies, semiespecies, especies).
A pesar de estos nuevos desarrollos, las comparaciones basadas en los métodos estándar siguen siendo valiosas si se reconocen sus limitaciones. D. silvestris y heteroneura son mucho más cercanas alozímicamente que cualquier otro par de especies de Drosophila examinadas hasta ahora, incluidos todos los pares de especies "hermanas", subespecies y semiespecies (Ayala et al. 1974b). De hecho, los coeficientes de similitud para las poblaciones dentro de silvestris o heteroneura son del mismo orden de magnitud que los entre las mismas dos especies. Las pequeñas diferencias que existen entre pares de poblaciones de las dos especies, además, no son las mismas en las tres áreas estudiadas; es decir, no involucran los mismos loci o alelos.
Como se discutió en la introducción, seguimos reconociendo estas dos entidades como buenas especies biológicas. Son morfológicamente distintas, ampliamente simpátricas, comportamentalmente aisladas y diferentes en sus polimorfismos cromosómicos. Las diferencias morfológicas dependen de aproximadamente catorce poligenes (Val 1976).
Una característica notable e importante de estas especies, sin embargo, es el hecho de que los híbridos recíprocos, obtenidos en el laboratorio, son completamente fértiles en ambos sexos (Craddock 1974; Ahearn y Val 1975). En consecuencia, se debe considerar la hibridación natural. Aunque Craddock (1974) no encontró evidencia de esto, Kaneshiro y Val (1977) han informado que aproximadamente el dos por ciento de las moscas salvajes capturadas en Kahuku Ranch pueden ser identificadas como híbridos por medios morfológicos. Esta es la misma región donde se encontraron híbridos naturales entre dos otras especies (Carson, Nair y Sene 1975). El análisis morfométrico de la forma de la cabeza se ha utilizado con éxito para especificar la parentela recíproca exacta de ciertos individuos híbridos F1. En un caso, una hembra D. silvestris capturada en la naturaleza produjo descendencia compuesta enteramente por híbridos F1. Dado que cada hembra salvaje fue aislada inmediatamente de todos los machos al ser capturada, está claro que los apareamientos relevantes tuvieron lugar en la naturaleza. Se debe enfatizar que el trasfondo de extensos estudios morfométricos de híbridos de laboratorio (Val 1976) ha preparado el escenario para estas nuevas observaciones naturales y, en consecuencia, el diagnóstico de estos casos se basa en evidencia comparativa sólida. Todos los individuos híbridos naturales han sido excluidos de los datos electroforéticos presentados en este documento.
Un hecho adicional que suma a la situación inusual respecto a estas especies es su estrecha similitud ecológica. Ambas han sido criadas a partir de los mismos tallos en descomposición del planta huésped común, Clermontia. Ambas, asimismo, forman sus leks en lugares muy similares, de modo que machos y hembras de ambas especies interactúan en la naturaleza.
¿Cuál es la razón de la gran similitud alozímica de estas especies? Se sugieren dos posibilidades. Primero, cuando ocurrió la divergencia entre las especies originalmente, puede haber estado acompañada de cierta cantidad de divergencia electroforética. Las similitudes observadas actualmente requerirían que tales diferencias fueran erosionadas por hibridación introgresiva posterior a la especiación. Una segunda opinión sostiene que solo un cambio electroforético menor acompañó los eventos de especiación en primer lugar. Esto podría estar correlacionado con la considerable cercanía ecológica.
Favoramos la segunda opinión por varias razones. Las dos especies son más similares en Olaa. Sin embargo, solo se ha obtenido evidencia sólida de hibridación reciente para Kahuku, donde las especies son alozímicamente más distantes. En Kahuku, además, las poblaciones naturales no muestran evidencia de formar un “enjambre híbrido” que se asemeje a la amplia recombinación genética observada en cruces de laboratorio. De hecho, la introgressión entre las especies probablemente está ocurriendo, pero puede estar restringida a un espectro genotípico estrecho por selección natural. Así, solo los genes de importancia biológica menor (el “sistema abierto”, Carson 1975) pueden fluir a través de la barrera de especies. Por el contrario, otros genes pueden marcar o ser una parte funcional de un sistema cerrado de balances epistáticos únicos de una u otra de las especies. La selección podría, por lo tanto, servir para inhibir la ruptura de tal “sistema cerrado” (Carson 1975).
Debido a la novedad geológica de la isla de Hawái, estamos tentados a sugerir que D. silvestris y D. heteroneura pueden ser realmente nuevas formaciones en el tiempo. Esta circunstancia podría ser la causa principal de la similitud electroforética de las dos especies. De hecho, pueden ser mucho más nuevas que los clásicos pares de especies "hermanas" (ver Carson 1976b). Al igual que en otras aparentes neoespecies, (por ejemplo Gottlieb 1974; Turner 1974; Avise, Smith y Ayala 1975), la diferenciación alozímica ha sido relativamente leve. De hecho, existe la posibilidad de que la diferencia alozímica simplemente pueda correlacionarse con el tiempo transcurrido desde el evento cladístico que separó las dos entidades comparadas (Avise, Smith y Ayala 1975; Carson 1976a). Las diferencias morfológicas, por otro lado, pueden servir como mejores indicadores de los efectos subyacentes de los genes reguladores que caracterizan las importantes diferencias genéticas entre las especies recién formadas.
Autores: F. M. Sene, H. L. Carson
Literatura citada:
- Ahearn, J. N., H. L. Carson, Th. Dobzhansky y K. Y. Kaneshiro, 1974 Aislamiento etológico entre tres especies del subgrupo planitibia de Drosophila hawaiana. Proc. Natl. Acad. Sci. U.S. 71: 901—903.
- Ahearn, J. N. y F. C. Val, 1975 Híbridos interespecíficos fértiles de dos Drosophila hawaianas simpátricas. Genetics 80: s9.
- Avise, J. C., J. J. Smith y F. J. Ayala, 1975 Diferenciación adaptativa con poco cambio génico entre dos peces nativos de California. Evolution '29: 411—426.
- AYALA, F. J., 1975 Diferenciación genética durante el proceso de especiación. Evolutionary Biology 8: 1—78.
- Ayala, F. J., J. R. Powell, M. L. Tracey, C. A. Mourão y S. Pérez-Salas, 1972 Variabilidad enzimática en el grupo Drosophila willistoni. IY Variación génica en poblaciones naturales de Drosophila willistoni. Genetics 70: 113—139.
- Ayala, F. J., M. L. Tracey, L. G. Barr y J. G. Ehrenfeld, l9f4a Diferenciación genética y reproductiva de la subespecie, Drosophila equinoxialis caribbensis. Evolution 28: 24—41.
- Ayala F. J., M. L. Tracey, D. Hedgecock y R. C. Richmond, 1974b Diferenciación genética durante el proceso de especiación en Drosophila. Evolution 28: 576—592.
- Carson, H. L. 1975 La genética de la especiación a nivel diploide. Am. Naturalist 109: 83—92, 1976a Inferencia del tiempo de origen de algunas especies de Drosophila. Nature 259: 395—396, 1976b Diferencias genéticas entre especies recién formadas. Bioscience 26: 700—701.
- Carson, H. L., P. S. Nair y F. M. Sene, 1975 Híbridos de Drosophila en la naturaleza: Prueba de intercambio genético entre especies simpátricas. Science 189: 806—807.
- Carson, H. L. y H. D. Stalker, 1968 Relaciones de cromosomas politénicos en especies hawaianas de Drosophila. II. El subgrupo D. planitibia. Univ. Texas Publ. 6818: 355-365.
- Coyne, J. A., 1976 Falta de similitud génica entre dos especies hermanas de Drosophila revelada por diversas técnicas. Genetics 84: 593—607.
- Craddock, E. M., 1974 Relaciones reproductivas entre especies homosecuenciales de Drosophila hawaiana. Evolution 28: 593—606.
- Craddock, E. M. y H. L. Carson, 1975 Variabilidad cromosómica en una especie endémica de Drosophila hawaiana. Genetics 80: s23.
- Gottlieb, L. D., 1974 Confirmación genética del origen de Clarkia lingulata. Evolution 2B: 244-250.
- Hardy, D. E., 1965 insectos de Hawái, Vol. 12. p. 814. University of Hawaii Press, Honolulu.
- Johnson, W. E. y H. L. Carson, 19f5 Variación alozímica en Drosophila silvestris. Genetics 80: s46.
- Johnson, W. E., H. L. Carson, K. Y. Kaneshiro, W. W. M. Steiner y M. M. Cooper, 1975 Variación genética en Drosophila hawaiana. II. Diferenciación alozímica en el subgrupo D. planitibia. pp. 563—584. En: Isoenzimas IV. Genética y Evolución. Editado por C. L. Markert. Academic Press, Nueva York.
- Kaneshiro, K. Y., 1976 Aislamiento etológico y filogenia en el subgrupo planitibia de Drosophila hawaiana. Evolution 30: 740-745.
- Kaneshiro, K. Y. y F. C. Val, l977 Hibridación natural entre un par simpátrico de Drosophila hawaiana. Am. Naturalist (en prensa).
- Lewontin, R. C. y J. L. Hubby, 1966 Un enfoque molecular para el estudio de la heterocigosis génica en poblaciones naturales. II. Cantidad de variación y grado de heterocigosis en poblaciones naturales de Drosophila pseudoobscura. Genetics 54: 595—609.
- Nei, M., 1972 Distancia genética entre poblaciones. Am. Naturalist 106: 283—292.
- Powell, I. R., 1975 Variación proteica en poblaciones naturales de animales. Evol. Biol. 8: 79-113.
- Simon, R. S., R. C. LEWONTin y A. A. Fzczou, 19 Z6 Heterogeneidad genética dentro de “alelos” electroforéticos de xantina deshidrogenasa en Drosophila pseudoobscura. Genetics 84: 609—629.
- Steiner, W. W. M. y W. E. Johnson, 1973 Técnicas para la electroforesis de Drosophila hawaiana. US/IBP Island Ecosystems IRP. Tech. Report 30: ii + 21 pp.
- Turner B. J., 1974 Divergencia genética de especies de peces de la cuenca de Death Valley: Evidencia bioquímica versus morfológica. Evolution fl8: 281-294.
- Val, F. C., 1976 Genética de las diferencias morfológicas entre dos especies de Drosophila interfértiles. Genetics 83: s78.
