Variabilidad del ADN satelital pBuM189 entre poblaciones sudamericanas de Drosophila buzzatii
Traducción automática
El artículo original está escrito en idioma EN (enlace para leerlo) .
El ADN satelital pBuM189 fue analizado en poblaciones de Drosophila buzzatii que cubren la mayor parte de la distribución de la especie en América del Sur. Este satDNA consiste en monómeros ricos en A+T de 189 pb y estudios previos mostraron una rápida tasa de cambio evolutivo de este componente del genoma de D. buzzatii. Se estudiaron un total de 63 unidades de repetición de pBuM189 de 14 poblaciones de D. buzzatii (9 de Brasil y 5 de Argentina). La variabilidad nucleotídica promedio entre las 63 repeticiones es del 4.2 %. Al menos una repetición (Juan/4) parece ser parte de otra subfamilia de satDNA pBuM189. La alineación nucleotídica de las 63 repeticiones reveló ninguna sustitución nucleotídica específica, o indels, que pudieran discriminar cada población o grupos de poblaciones geográficamente cercanas. Esta falta de diferenciación interpopulacional de satDNA es congruente con datos previos de mtDNA que indican un alto flujo génico y muy poca diferenciación poblacional a lo largo de la mayor parte de la distribución de D. buzzatii en América del Sur. El flujo génico podría haber sido posible durante eventos de glaciación en el Pleistoceno, como el que ocurrió entre hace 13,000 y 18,000 años, cuando D. buzzatii probablemente tenía una distribución más continua de lo que se observa hoy en día.
La mosca cactofílica Drosophila buzzatii (buzzatii cluster, repleta group) tiene una amplia distribución en América del Sur, encontrándose en la mayoría de los territorios de Argentina, Bolivia, Paraguay y Brasil. La especie utiliza tejidos en descomposición de varios cactus de pera espinosa (Opuntia ssp.) y cactus columnares para alimentarse y reproducirse (PEREIRA et al. 1983). Durante los últimos doscientos años, los cactus Opuntia han sido transportados por el hombre a varias regiones del mundo, incluidos países de la cuenca mediterránea, África y Australia. En consecuencia, D. buzzatii migró con sus huéspedes. Esta situación hizo que D. buzzatii fuera un organismo apropiado para abordar preguntas relacionadas con los cambios genéticos durante el proceso de colonización (FONTDEVILA et al. 1982; HALLIBURTON y BARKER 1993; ROSSI et al. 1996; FRYDENBERG et al. 2002).
Según los datos de inversión cromosómica, el Chaco argentino es el centro más probable de su radiación (WASSERMAN 1962; CARSON y WASSERMAN 1965). D. buzzatii también es muy abundante en el Chaco, asociado con varias especies diferentes de cactus (Vilela et al. 1980). En general, las poblaciones brasileñas están distribuidas de manera discontinua a lo largo de pequeñas áreas de vegetación xerofítica, donde se han encontrado en frecuencias muy bajas (VILELA et al. 1983; TIDON-SKLORZ et al. 1994; TIDON-SKLORZ y SENE 1995). La única excepción es el sur de Brasil, donde se encontraron poblaciones relativamente grandes (VILELA et al. 1983; RUIZ et al. 2000).
Se ha postulado que D. buzzatii colonizó Brasil a través de cambios en la distribución de la vegetación árida durante eventos de glaciación, como los que ocurrieron a finales del Pleistoceno (BARKER et al. 1985; FIGUEIREDO y SENE 1992).
BAIMAI et al. (1983) analizaron los cromosomas en metafase de varias poblaciones de D. buzzatii de Argentina y Brasil. Todas las poblaciones muestreadas exhiben la misma placa metafásica. BARKER et al. (1985) no encontraron diferencias alozimáticas significativas entre las poblaciones sudamericanas. Solo una pequeña diferenciación local parece haber ocurrido en las poblaciones brasileñas de Bahia (noreste) y Tramanda´1 (sur). En otro estudio, FIGUEIREDO y SENE (1992) informaron que de 16 inversiones cromosómicas detectadas en Argentina, solo dos estaban presentes en las poblaciones brasileñas y ninguna se encontró en el noreste de Brasil. ROSSI et al. (1996) y DE BRITO et al. (2002) encontraron altos niveles de uniformidad de mtDNA entre las poblaciones sudamericanas de D. buzzatii y varias pruebas indicaron un alto flujo genético a lo largo de la mayor parte de la distribución geográfica de D. buzzatii . DE BRITO et al. (2002) también calcularon que D. buzzatii ha estado presente en Brasil durante al menos 100 mil años, sugiriendo una expansión preholocena de las poblaciones argentinas de D. buzzatii hacia Brasil.
Con el fin de obtener más información sobre la variabilidad genética de D. buzzatii en América del Sur, buscamos un ADN nuclear. El ADN satelital (satDNA) es una clase de ADN no codificante altamente repetitivo que se organiza en tandas en grandes arreglos homogéneos en el genoma de casi todos los organismos eucariotas. La rápida tasa de cambio evolutivo de las secuencias de satDNA está respaldada por varias familias de satDNA que resultaron ser específicas de especie (BACH-MANN et al. 1989; KING y CUMMINGS 1997).
Estudios previos mostraron que las secuencias de satDNA podrían ser útiles en la discriminación de poblaciones conspecíficas (ELDER y TURNER 1994; WU et al. 1999).
KUHN et al. (1999) describieron el ADN satelital pBuM189 de D. buzzatii. Consiste en unidades de repetición ligeramente ricas en AT y de 189 pb de longitud. Las cinco secuencias pBuM189 descritas se obtuvieron del ADN genómico de solo una población de D. buzzatii. Los experimentos de hibridación y PCR no lograron detectar secuencias pBuM189 en el genoma de especies estrechamente relacionadas como D. serido, D. borborema o D. koepferae. Por lo tanto, los datos disponibles apuntan a una rápida tasa de cambio evolutivo de este componente del ADN nuclear de D. buzzatii.
En el presente estudio, analizamos 63 secuencias de pBuM189 de cepas que representan 14 poblaciones de D. buzzatii (9 brasileñas y 5 argentinas). Las poblaciones cubren la mayor parte de la distribución geográfica de esta especie en América del Sur.
Materiales y métodos
Muestras
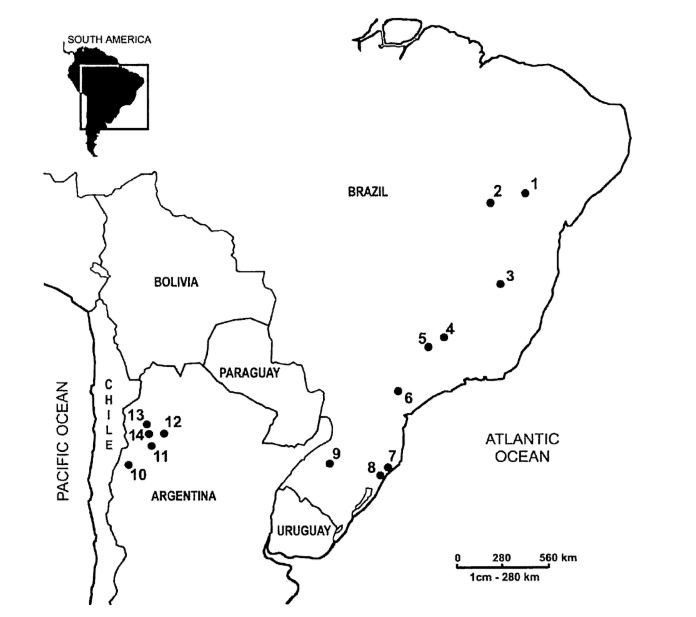
La ubicación geográfica de todas las poblaciones de D. buzzatii muestreadas se muestra en la Fig. 1. Las cepas utilizadas con sus respectivas localidades son: D69R5 (1); J79H41 (2); D54F5 (3); H86G8 (4); D42F2 (5); H99X6 (6); H42F1 (7); J25A20 (8); J28E15 (9); ArgE3 (10); Cat (11), Hondo (12); Salta (13) y Ticucho (14).
Técnicas moleculares
Las secuencias de pBuM189 fueron aisladas originalmente del genoma de D. buzzatii después de la digestión de ADN con la enzima MspI (KUHN et al. 1999). En el presente trabajo, se utilizó la enzima SspI (que también está presente solo una vez por unidad de repetición de pBuM189) para facilitar el procedimiento de clonación. Los fragmentos de satDNA pBuM189 fueron identificados por su apariencia como bandas prominentes de aproximadamente 190 pb en la mancha de fondo del ADN genómico. El ADN de la banda de ADN de 190 pb fue eluido del gel mediante incubación nocturna en 500mM NaAc; 1mM EDTA. Los fragmentos recuperados fueron ligadas en un vector de plásmido pUC18 y transformados en células competentes de Escherichia coli DH5-a. Los clones recombinantes fueron seleccionados como colonias blancas en placas de ampicilina que contenían X-gal (5-bromo-4-cloro-3-indolil-β-D-galactósido) e IPTG (isopropilo-β-D-tiogalatopiranósido). La reacción de ADN plantilla para la secuenciación se preparó de acuerdo con el manual del BigDye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). La secuenciación automática de ADN se realizó en un secuenciador ABI Prism™ 377 (Perkin-Elmer).
El programa MEGA 2.1 (KUMAR et al. 2001) se utilizó para la estimación de la variabilidad de nucleótidos, distancias genéticas y la construcción de un dendrograma NJ.
Resultados y discusión
Como ya se esperaba a partir de los datos de secuencia de pBuM189 previamente, la digestión de ADN genómico de 13 D. buzzatii cepas con SspI produjo una banda de ADN fuertemente teñida de aproximadamente 190 pb, correspondiente a los monómeros de pBuM189, seguida de bandas de señales de intensidad decreciente de 380 pb (dímeros), 570 pb (trímeros), y así sucesivamente. La banda de ADN de 190 pb de cada digestión se eluyó independientemente del gel y se clonó. Un total de 58 clones fueron secuenciados, 39 de 8 poblaciones brasileñas (localidades 1 – 4, 6 – 9) y 19 de 5 poblaciones argentinas (localidades 10 – 14). La alineación de nucleótidos de estos 58 clones con los 5 repeticiones de pBuM189 (sert/1 – 5 en el presente trabajo) de la localidad (5) descrita anteriormente por KUHN et al. (1999) demostró que todos representan monómeros de satDNA de pBuM189 (Fig. 2).

Los 63 repeticiones pBuM189 son ligeramente ricas en A+T (71 %) y la variabilidad promedio de la secuencia es del 4.2 %. La principal fuente de variación entre las 63 repeticiones pBuM189 son las sustituciones de nucleótidos individuales, siendo el 69 % específicas de la repetición y el resto comunes a dos o más repeticiones. Solo se encontraron 11 indels.
Una repetición pBuM189 parece ser excepcional. El clon Juan/4 de la localidad (10) mostró niveles inesperadamente altos de divergencia de secuencia en todas las comparaciones por pares (22.1 % en promedio y siempre superando el 19 %). Además, mientras que las secuencias típicas de pBuM189 presentan en promedio un 1.7 % de sustituciones de nucleótidos específicas de la repetición, se encontraron 30 en la repetición Juan/4 (por ejemplo, T en las posiciones 11 y 42). Tal alto grado de divergencia de nucleótidos sugiere que la repetición Juan/4 podría pertenecer a otra subfamilia de pBuM189. Desde un punto de vista teórico, el origen de las subfamilias de satDNA depende de variables como el número de copias, la distribución genómica de la familia de satDNA y las tasas relativas de intercambios no recíprocos dentro y entre cromosomas no homólogos (DOVER 1986).
Si se excluye el repetido Juan/4 de la muestra, la variabilidad nucleotídica promedio de las 62 secuencias restantes de pBuM189 disminuye al 3.7 %. Este valor es casi idéntico al 3.8 % calculado por KUHN et al. (1999) basado en cinco secuencias de pBuM189 aisladas de solo una población de D. buzzatii (localidad 5).
Entre los otros repeticiones de pBuM189, Ticu/4 de la localidad (14) es la más similar a Juan/4 por presentar dos nucleótidos específicos (G en la posición 70 y A en la posición 88) y un indel (posición 49) que son exclusivos del repetido Juan/4 y varias otras sustituciones en común con Juan/4 y otros repeticiones (por ejemplo, T en la posición 36 o A en la posición 133). El repetido Ticu/4 en sí es el repetido de pBuM189 más divergente (10.2 % en promedio) después de Juan/4, presentando 6 sustituciones nucleotídicas específicas del repetido (posiciones 1, 69, 79, 97, 98, 167 y 188). Por lo tanto, el Ticu/4 debe considerarse como un paso evolutivo intermedio entre los repeticiones "típicos" de pBuM189 y el altamente divergente repetido Juan/4. Curiosamente, ambos repeticiones raras Juan/4 y Ticu/4 son de Argentina, donde la especie probablemente se originó (WASSERMAN 1962; CARSON y WASSERMAN 1965).
Según datos teóricos y empíricos, la diferenciación de las secuencias de satDNA debería surgir de acuerdo con el concepto de evolución concertada (DOVER 1986; ELDER y TURNER 1994). En otras palabras, los arreglos de satDNA de diferentes poblaciones o especies podrían ser homogeneizados por diferentes mutaciones debido al efecto de mecanismos moleculares como el cruce desigual y la conversión de genes acoplados a la aislamiento genético y la deriva genética.
La alineación de nucleótidos de los 63 repeticiones de pBuM189 de 14 poblaciones de D. buzzatii (4.5 % de repeticiones en promedio por población) no reveló ninguna sustitución de nucleótido específica, o indel, que pudiera discriminar una población específica o grupos de poblaciones geográficamente cercanas (Fig. 2). En algunos casos, más de dos clones compartieron una secuencia de nucleótidos idéntica, como en el caso de los clones Capao/3, Grao/3, M.Cha/6 y Catam/1 o M.Cha/1 y Santi/4, pero provienen de localidades geográficas bastante distantes. Por otro lado, no se encontraron repeticiones idénticas en una sola población.
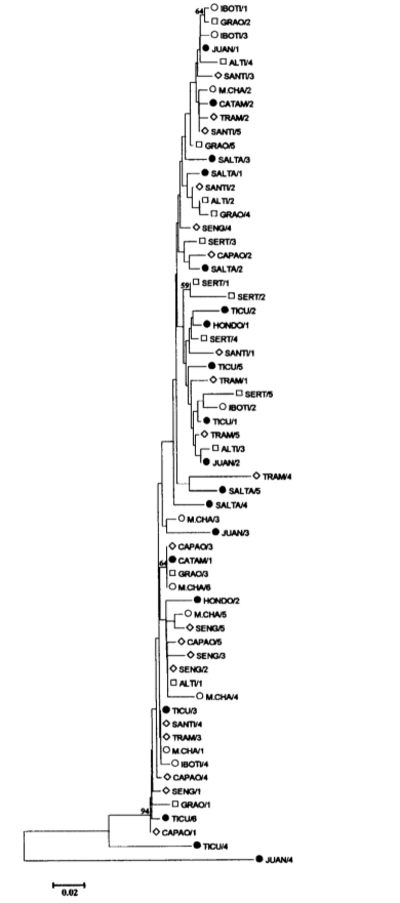
Las distancias genéticas entre las 63 secuencias nucleotídicas de pBuM189 se calcularon de acuerdo con el método de dos parámetros de Kimura (KIMURA 1980) y se construyó un dendrograma de "vecinos más cercanos" (SAITOU y NEI 1987) (Fig. 3). La secuencia Juan/4 se ubicó en una rama claramente separada de los otros repeticiones de pBuM189, apoyando la suposición de que pertenece a otra subfamilia de satDNA de pBuM189. La repetición Ticu/4 se ubicó en una rama entre la Juan/4 y las restantes 61 repeticiones de pBuM189. Dentro de las restantes 61 repeticiones de pBuM189, no se pudo observar ningún clúster que contenga secuencias de pBuM189 de una población o grupos de poblaciones geográficamente cercanas.
En conjunto, los datos indican altos niveles de similitud en la secuencia pBuM189 entre las poblaciones de D. buzzatii que se encuentran, en algunos casos, a más de 3000 km de distancia. Tal falta de diferenciación interpopulacional de satDNA es congruente con los datos de mtDNA que indican un alto flujo génico y muy poca subestructura poblacional a lo largo de la mayor parte de la distribución de D. buzzatii en América del Sur (ROSSI et al. 1996; DE BRITO et al. 2002). En consecuencia, debe haber una explicación adaptativa para la pérdida de polimorfismo de inversión cromosómica en las poblaciones brasileñas (en comparación con las poblaciones argentinas), reportada por FIGUEIREDO y SENE (1992).
Actualmente, el Chaco (norte de Argentina y este de Bolivia) y la Caatinga (noreste de Brasil) son las únicas dos áreas extensas de vegetación árida en América del Sur, presentando una alta densidad y diversidad de especies de cactus. En otras áreas, las cactáceas son menos frecuentes y están distribuidas de manera discontinua. Lo mismo ocurre con las poblaciones de D. buzzatii , que dependen de los cactus para alimentarse y reproducirse. Se acepta ampliamente que el clima global experimentó ciclos recurrentes de clima frío-seco y cálido-húmedo durante el Pleistoceno, cuando ocurrieron al menos cuatro eventos glaciares importantes y muchos menores. Por ejemplo, en el último evento de glaciación (hace 18,000 a 13,000 años), el clima en América del Sur era seco y el Chaco estaba conectado a las Caatingas a través de formaciones xerofíticas en el centro de Brasil y la costa atlántica (AB'SABER 1977). En consecuencia, D. buzzatii probablemente tuvo una distribución mucho más continua de lo que se observa hoy. Tal situación podría haber permitido el flujo génico entre las poblaciones de D. buzzatii en América del Sur en un gran grado.
Hasta la fecha, la mayoría de los DNAs satélites descritos en la literatura fueron aislados de individuos de una sola cepa o localidad. Los datos presentes son uno de los pocos ejemplos que proporcionan información sobre la variabilidad de la secuencia de satDNA entre poblaciones naturales de una especie ampliamente distribuida.
Autores: Gustavo C. S. Kuhn, Fernando F. Franco, Wilson A. Silva Jr, Nilce M. Martinez-Rossi y Fabio M. Sene
Referencias:
- Ab'Saber, N. A. 1977. Espacios ocupados por la expansión de los climas secos de América del Sur, por ocasión de los períodos glaciares cuaternarios. – Paleoclimas 3: 1 – 19.
- Bachmann, L., Raab, M. y Sperlich, D. 1989. ADN satélite y especiación: un ADN satélite específico de especie de Drosophila guanche. – Z. Zool. Syst. Evol. Forsch. 27: 84 – 93.
- Baimai, V., Sene, F. M. y Pereira, M. A. Q. R. 1983. Heterocromatina y diferenciación cariotípica de algunas especies de cactus de cría neotropical del grupo Drosophila repleta . – Genetica 67: 81 – 92.
- Barker, J. S. F., Sene, F. M., East, P. P. et al. 1985. Polimorfismo alozimático y cromosómico de Drosophila buzzatii en Brasil y Argentina. – Genetica 67: 161 – 170.
- Carson, H. L. y Wasserman, M. 1965. Un polimorfismo cromosómico ampliamente distribuido en una especie ampliamente distribuida, Drosophila buzzatii. – Am. Nat. 99: 111 – 115.
- De Brito, R. A., Manfrin, M. H. y Sene, F. M. 2002. Filogeografía del ADN mitocondrial de poblaciones brasileñas de Drosophila buzzatii. – Genet. Mol. Biol. 2: 161 – 171.
- Dover, G. A. 1986. Impulso molecular en familias multigénicas: cómo surgen, se propagan y son asimiladas las novedades biológicas. – Trends Genet. 2: 159 – 165.
- Elder, F. J. y Turner, B. J. 1994. Evolución concertada a nivel poblacional: secuencias de ADN satélite HindIII de peces de alevines. – Proc. Natl Acad. Sci. 91: 994 – 998.
- Figueiredo, V. L. C. y Sene, F. M. 1992. Variabilidad cromosómica en poblaciones brasileñas de Drosophila buzzatii (Diptera, Drosophilidae). – Revta. Bras. Biol. 52: 555 – 561.
- Fontdevila, A., Ruiz, A., Ocana, J. et al. 1982. La historia evolutiva de Drosophila buzzatii. II. ¿Cuánto ha cambiado el polimorfismo cromosómico en la colonización? – Evolution 36: 843 – 851.
- Frydenberg, J., Pertoldi, C., Dahlgaard, J. et al. 2002. Variación genética en poblaciones originales y colonizadoras de Drosophila buzzatii analizadas por loci de microsatélites aislados con un nuevo método de cribado PCR. – Mol. Ecol. 11: 181 – 190.
- Halliburton, R. y Barker, J. S. F. 1993. Falta de variación en el ADN mitocondrial en Drosophila buzzatii australiano. – Mol. Biol. Evol. 10: 484 – 487.
- Kimura, M. 1980. Un único método para estimar la tasa evolutiva de sustituciones de bases a través de estudios comparativos de secuencias de nucleótidos. – J. Mol. Evol. 16: 111 – 120.
- King, L. M. y Cummings, M. P. 1997. La variación de la secuencia de repetición de ADN satélite es baja en tres especies de los escarabajos enterradores del género Nicrophorus (Coleoptera: Silphidae). – Mol. Biol. Evol. 14: 1088 – 1095.
- Kuhn, G. C. S., Bollgönn, S., Sperlich, D. et al. 1999. Caracterización de un ADN satélite específico de especie de Drosophila buzzatii. – J. Zool. Syst. Evol. Res. 37: 109 – 112.
- Kumar, S., Tamura, K., Jakobsen, I. B. et al. 2001. MEGA2: software de análisis de genética evolutiva molecular. – Arizona State Univ.
- Pereira, M. A. Q. R., Vilela, C. R. y Sene, F. M. 1983. Notas sobre sitios de cría y alimentación de algunas especies del grupo repleta del género Drosophila (Diptera, Drosophilidae). – Ciência e Cultura 35: 1313 – 1319.
- Rossi, M.S., Barrio, E., Latorre, A. et al. 1996. La historia evolutiva de Drosophila buzzatii. XXX. Polimorfismo del ADN mitocondrial en poblaciones originales y colonizadoras. – Mol. Biol. Evol. 13: 314 – 323.
- Ruiz, A., Cansian, A. M., Kuhn, G. C. S. et al. 2000. El rompecabezas de especiación de Drosophila serido : uniendo nuevas piezas. – Genetica 108: 217 – 227.
- Saitou, N. y Nei, M. 1987. El método de vecino-uniendo: un nuevo método para reconstruir árboles filogenéticos. – Mol. Biol. Evol. 4: 406 – 425.
- Tidon-Sklorz, R. y Sene, F. M. 1995. Fauna de Drosophila (Diptera, Drosophilidae) en el área norte de la ‘‘Cadeia do Espinhac¸o'', estados de Minas Gerais y Bahia, Brasil: aspectos biogeográficos y ecológicos. – Iheringia Ser. Zool. 78: 85 – 94.
- Tidon-Sklorz, R., Vilela, C. R., Sene, F. M. et al. 1994. El género Drosophila (Diptera, Drosophilidae) en la Serra do Cipó, estado de Minas Gerais, Brasil. – Revta. Bras. Ent. 38: 627 – 637.
- Vilela, C. R., Sene, F. M. y Pereira, M. A. Q. R. 1980. Sobre la fauna de Drosophila del Chaco y las laderas orientales de los Andes en Argentina. – Revta. Bras. Biol. 40: 837 – 841. Vilela, C. R., Pereira, M. A. Q. R. y Sene, F. M. 1983. Datos preliminares sobre la distribución geográfica de especies de Drosophila dentro de los dominios morfoclimáticos de Brasil. II. El grupo repleta . – Ciên. Cult. São Paulo 35: 66 – 70.
- Wasserman, M. 1962. Estudios citológicos del grupo repleta del género Drosophila. V. El subgrupo mulleri . – Univ. Texas Publ. 6205: 85 – 118.
- Wu, W. L., Wang, J. P., Tseng, M. C. et al. 1999. Clonación y variabilidad genética de un ADN repetitivo HindIII en Acrossocheilus paradoxus (Cyprinidae). – Genome 42: 780 – 788.

/public-service/media/default/428/W1Mkp_671a13a60217a.png)
/public-service/media/default/434/FFP3Z_671a14a5b5af8.jpeg)
/public-service/media/default/433/0dO9S_671a149056e56.jpeg)